|
使用 CUT&RUN 與 CUT&Tag 技術解決傳統 ChIP 痛點 CUT&RUN (Cleavage Under Targets and Release Using Nuclease) 與 CUT&Tag (Cleavage Under Targets and Tagmentation) 都是由美國福瑞德哈金森癌症研究中心的 Henikoff 實驗室所開發,其中 CUT&Tag 可視為是 CUT&RUN 的升級版。這兩項技術都可以取代傳統染色質免疫沉澱 (chromatin immunoprecipitation, ChIP),協助研究人員以更快速簡便的方式與更少量的樣本探討細胞內蛋白質與基因體 DNA 的交互作用,並獲取更精準的實驗結果 [1]。以下就為您介紹 CUT&RUN 與 CUT&Tag 技術原理以及通過 CUT&RUN 與 CUT&Tag 實驗確校的抗體產品。
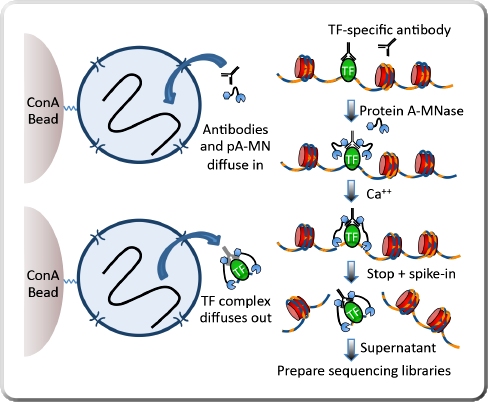
CUT&RUN CUT&RUN 是 Henikoff 實驗室基於 ChIC (chromatin immunocleavage) 技術改良而成。在 ChIC 技術中,樣本不需要經過超音波破碎,而是透過將微球菌核酸酶 (micrococcal nuclease, MNase) 引導至目標蛋白質處(例如:組蛋白 (histones) 或轉錄因子 (transcription factors, TF)),使其切割下該區域的染色質片段。ChIC 技術使用了目標蛋白質專一性抗體以及融合有 Protein A 的 MNase(簡稱 pA-MNase 或 pA-MN),藉由 Protein A 能夠與 IgG 抗體結合的特性來引導 pA-MNase 至目標蛋白質處,達到定位切割效果 [2]。 CUT&RUN 技術則是進一步採用包覆有凝集素 (lectin) 「刀豆球蛋白 A (concanavalin A, Con A)」的磁珠來提高實驗效率。在進行 CUT&RUN 實驗時,樣本不再需要經過交聯固定與超音波破碎處理。Con A 磁珠可以透過與細胞膜或細胞核膜上的醣蛋白結合,直接將樣本抓取固定於磁珠上。目標蛋白質專一性抗體和 pA-MNase 會經由核孔進入細胞核中,並在鈣離子的活化下完成切割反應。在加入螯合劑終止酵素反應後,通過簡單離心步驟即可將切割下來的「pA-MNase:抗體:目標蛋白質:DNA 結合片段」複合物分離於上清液中,準備進行 DNA 純化與定序分析 [3]。 CUT&RUN 技術原理。 IMAGE © Elife. 2017 Jan 16;6:e21856. Fig. 1A [3]. CUT&RUN 技術公開後迅速獲得廣大迴響,Henikoff 實驗室曾在論文中寫道:「...since publication of our eLife paper we have distributed materials to >600 laboratories world-wide...」可見一斑 [4]。同時 CUT&RUN 技術也不斷地在精進改良,例如減少樣本量,甚至可達到單細胞等級;或是改使用同時融合有 Protein A 與 Protein G 的 MNase(簡稱 pAG-MNase),以改善對鼠類抗體結合效率不佳的問題 [4-6]。總結來說,我們可以將 CUT&RUN 技術優於 ChIP 的地方歸納為以下幾點:
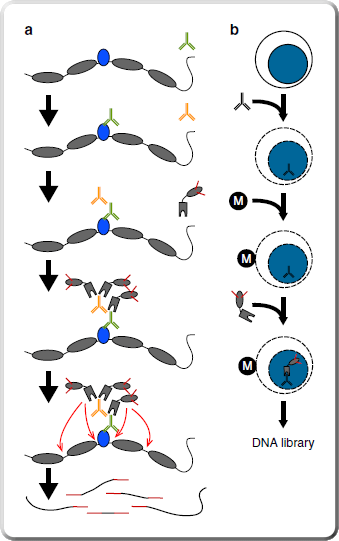
CUT&Tag CUT&Tag 則是將原本 CUT&RUN 技術中所使用的「MNase」替換成「Tn5 轉位酶 (Tn5 transposase)」,該轉位酶同樣會與 Protein A 融合,形成 pA-Tn5。pA-Tn5 同樣會透過目標蛋白質專一性抗體結合到目標染色質區域,並在鎂離子的活化下對目標染色質片段進行切割,同時將所帶有的定序建庫用標籤序列 (adapter) 銜接至 DNA 片段兩端。 CUT&Tag 技術原理。 IMAGE © Nat Commun. 2019 Apr 29;10(1):1930. Fig. 1 [7]. 相較於 CUT&RUN,CUT&Tag 技術流程更加快速,從細胞至取得適用於定序的 PCR 擴增序列庫最快一天即可完成,且樣本需求量可減少至 60 個細胞,甚至是單細胞。從定序結果來看,CUT&Tag-seq 的背景值比 CUT&RUN-seq 更低,具有更為出色的訊噪比,因此更加適合少量細胞或單細胞分析使用 [7]。目前 CUT&Tag 技術已衍生出多項適用於單細胞與多體學 (Multi-omics) 研究的技術,感興趣者可前往 Henikoff 實驗室網頁了解相關資訊 [1]。
為了幫助科研人員更快取得研究成果,Abcam 以近乎苛求的品質標準打造 CUT&RUN 與 CUT&Tag 實驗用抗體,其設計核心包含:
使用 Abcam CUT&RUN 驗證抗體 Recombinant Anti-CTCF antibody [EPR18253] - ChIP Grade (ab188408) 進行 CUT&RUN-seq 與 ChIP-seq 實驗,結果顯示兩者數據有高度一致性。 CUT&RUN was performed using the pAG-MNAse (ab285373), 10⁵ HeLa cells and 5 µg of ab188408 [EPR18253]. The resulting DNA was sequenced on the Illumina NovaSeq 6000 to a depth of 10 million reads. The ChIP data was conducted on chromatin prepared from HeLa cells. Cells were fixed with 1% formaldehyde for 10 minutes. ChIP was performed with 10⁷ HeLa cells and 4 µg of ab188408 [EPR18253]. ChIP DNA was sequenced on the Illumina NovaSeq 6000 to a depth of 30 million reads. IMAGE © Abcam. 使用 Abcam CUT&RUN 驗證抗體 Recombinant Anti-Histone H3 (tri methyl K4) antibody [EPR20551-225] - ChIP Grade (ab213224) 進行 CUT&RUN-seq 與 ChIP-seq 實驗,結果顯示兩者數據有高度一致性。 CUT&RUN was performed using the pAG-MNAse (ab285373), 10⁵ HeLa cells and 2 µg of ab213224 [EPR20551-225]. The resulting DNA was sequenced on the Illumina NovaSeq 6000 to a depth of 10 million reads. The ChIP data was conducted on chromatin prepared from HeLa cells. Cells were fixed with 1% formaldehyde for 10 minutes. ChIP was performed with 10⁷ HeLa cells and 4 µg of ab213224 [EPR20551-225]. ChIP DNA was sequenced on the Illumina NovaSeq 6000 to a depth of 30 million reads. IMAGE © Abcam. CUT&RUN 抗體熱銷產品
CUT&Tag 抗體熱銷產品
最新優惠活動與完整產品資訊,歡迎洽詢 Abcam 台灣代理伯森生技。您可透過下方連結瀏覽更多相關資訊:
References
|
|||||||||||||||||||||||||||||||||||||||||||||||||||
 |
 |
||||||||||||||||||||||||||||||||||||||||||||||||||
伯森生物科技(股)公司 Blossom Biotechnologies, Inc.
網址 www.blossombio.com 客服 0800-059668
[ 📝 線上留言諮詢 ] [ ☎ 伯森業務專員聯絡資訊 ]
![使用 Abcam CUT&RUN 驗證抗體 Recombinant Anti-CTCF antibody [EPR18253] - ChIP Grade (ab188408) 進行 CUT&RUN-seq 與 ChIP-seq 實驗,結果顯示兩者數據有高度一致性](img/ab188408.png)
![使用 Abcam CUT&RUN 驗證抗體 Recombinant Anti-Histone H3 (tri methyl K4) antibody [EPR20551-225] - ChIP Grade (ab213224) 進行 CUT&RUN-seq 與 ChIP-seq 實驗,結果顯示兩者數據有高度一致性](img/ab213224.png)